就只看了你一眼 就已确定了来源
不同类型细胞在制备、扩增及修饰等体外操作过程中,经常会发生细胞间交叉污染。有效判断细胞的种属来源,和确保细胞在制备过程中无不同种属间细胞交叉污染,是细胞质量控制中的重要环节之一。
《中国药典》生物制品生产检定用动物细胞基质制备及质量控制规程中,明确“新建细胞系/株、细胞库 (MCB和WCB) 和生产终末细胞应进行鉴别试验,以确认为本细胞,且无其他细胞的交叉污染。细胞鉴别试验方法有多种,包括细胞形态、生物化学法(如同工酶试验)、免疫学检测(如组织相容性抗原、种特异性免疫血清)、细胞遗传学检测(如染色体核型、标记染色体检测)、遗传标志检测 [如DNA指纹图谱,包括短串联重复序列(STR)、限制片段长度多态性(RFLP-PCR)和内含子多态性(EPIC-PCR)法等] 以及其他方法(如杂交法、PCR法、报告基因法等)。应至少选择上述一种或几种方法对细胞进行种属和细胞株间及专属特性的鉴别。”
多种基于蛋白和DNA的方法可以用于动物物种鉴定,这些方法在实用性和所具有优势方面存在差异,也各有局限。国标 GB/T 40172-2021《哺乳动物细胞交叉污染检测方法通用指南》文件中对常用检测方法的原理、依据、局限和结果判读有原则性的描述。
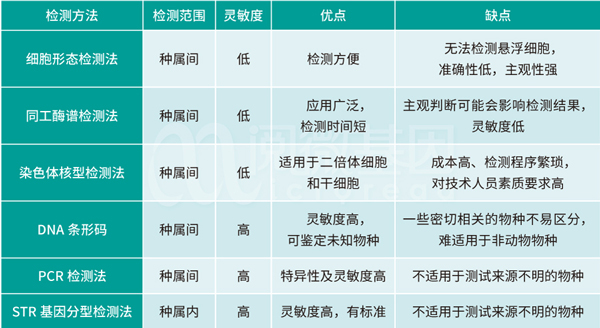
表1 细胞的身份鉴别常用检测方法对比
与基于蛋白质的技术相比,基于DNA的检测方法在进行物种鉴定时具有许多优势:
1、DNA比蛋白质更稳定,能够对无活力或高度降解的样本进行物种鉴定;
2、DNA检测技术方法往往比蛋白方法更快捷、便宜和灵敏;
3、基于PCR的检测方法易于标准化和数据化,减少人工判读的主观性,易于自动化。
因此,PCR检测法在物种鉴定中变得越来越普遍[1-6]。利用种属间保守基因(常用线粒体基因CO1或CYTB)中的差异处设计特异性引物,PCR扩增出目的产物后,通过琼脂糖凝胶电泳区分,判定是否检测到目标物种以及是否存在种属间交叉污染。
阅微基因的细胞种属污染鉴定项目已为上百家客户提供过检测服务,原针对11个生物实验室常用细胞种属(狗,非洲绿猴,人,牛,兔,猪,猫,大鼠,小鼠,叙利亚仓鼠,中国仓鼠)的多个基因设计PCR特异性引物,各物种单独扩增后琼脂糖凝胶电泳。随着客户需求以及对项目质量要求的增加,现依据国内外标准[7,8],完成项目升级及相应验证工作:
一、种属由原11个种属增加至15个(新增猕猴、马、绵羊、山羊);
二、针对线粒体COI基因,重新设计了片段长度有区别的单管混扩引物组;
三、在毛细管电泳平台实现数据分析及对种属污染的可视化鉴定;
四、试剂盒验证完善并提供质控品,在自己实验室也能轻松操作。

图1 检测到内参峰及猫物种识别峰,待检样本来源于猫
新产品有多好用呢?15个物种在单管内完成扩增,结果图谱一眼识别物种来源。你看,图1样本只有内参及猫物种出峰,其他物种不出峰,这个样本妥妥的是猫源细胞。
与传统PCR+跑胶方法相比,大大简化了实验流程,缩短了检测时间,再现性强,结果准确可靠。单物种检出限0.1ng/μL,污染识别率达1%,验证结果表明,阅微基因的细胞种属污染鉴定(多重PCR-CE法)检验结果准确可靠。
准确性
扩增15个物种的单一细胞系DNA时,内参峰均正常,物种识别峰正确且无非特异峰。饱和上样量条件下,仅目标物种出峰,近缘物种不出峰。证明该体系准确性与特异性良好。
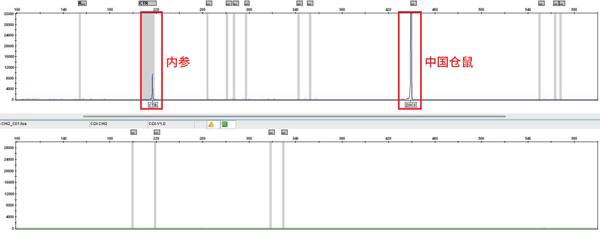
图2 中国仓鼠DNA饱和上样量检测结果图:仅中国仓鼠出峰,近缘物种不出峰
灵敏度
将15个物种细胞系DNA两两组合并按照1%比例进行混合,综合统计所有组合的污染物种峰高/主物种峰高比(见表2),结果证明该体系可以明确鉴定出1%含量的物种(检测范围内)污染。
表2 污染物种峰高/主物种峰高比统计

重复性
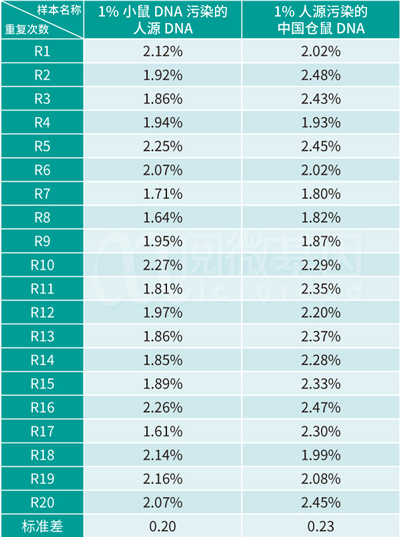
重复检测1%小鼠DNA污染的人源DNA和1%人源污染的中国仓鼠DNA 样本20次,污染物种峰高/主物种峰高比数据(见表3),结果显示体系100%可鉴定出污染种属情况(20/20)。
表3 污染物种峰高/主物种峰高比统计
作为在国内推出细胞鉴定服务的公司之一,阅微基因已在细胞身份鉴别项目积累了丰富的经验。除种属污染鉴定产品外,还可提供细胞形态、染色体核型分析、STR检测、DNA条形码体系开发等其他细胞身份鉴别检测服务。
现已为超过30,000例细胞发出身份证明,包含180多不同细胞系。客户覆盖国内知名疫苗企业、细胞公司、细胞治疗企业、生物药企及科研单位,并与国内各大细胞库有深刻且密切的合作。
未来我们也将持续优化产品,坚持为客户提供专业、可靠的检测服务,为客户的的产品申报和生产研发提供强有力的支持。
[1] Stacey, G.N., et al., Authentication of animal cell cultures by direct visualization of repetitive DNA, aldolase gene PCR and isoenzyme analysis. Biologicals, 1997. 25(1): p. 75-85.
[2] Parodi, B., et al., Species identification and confirmation of human and animal cell lines: a PCR-based method. Biotechniques, 2002. 32(2): p. 432-4, 436, 438-40.
[3] Liu, M.Y., et al., Identification and authentication of animal cell culture by polymerase chain reaction amplification and DNA sequencing. In Vitro Cell Dev Biol Anim, 2003. 39(10): p. 424-7.
[4] Lopez-Andreo, M., et al., Identification and quantitation of species in complex DNA mixtures by real-time polymerase chain reaction. Anal Biochem, 2005. 339(1): p. 73-82.
[5] Steube, K.G., A.L. Koelz, and H.G. Drexler, Identification and verification of rodent cell lines by polymerase chain reaction. Cytotechnology, 2008. 56(1): p. 49-56.
[6] Losi, C.G., et al., An alternative method to isoenzyme profile for cell line identification and interspecies cross-contaminations: cytochrome b PCR-RLFP analysis. In Vitro Cell Dev Biol Anim, 2008. 44(8-9): p. 321-9.
[7] GB/T 40172-2021 哺乳动物细胞交叉污染检测方法通用指南
[8] ANSI/ATCC ASN-0003-2015







